基因工厂云探秘系列2--免费序列优化,提升载体构建成功率 解锁高效基因表达
北京2024年9月12日 /美通社/ -- 载体构建是蛋白表达等很多实验的起点,也许你知道可以通过优化启动子、调整表达载体、选择合适的宿主细胞、控制培养条件等来提高蛋白表达量,但你是否关注过密码子优化这个关键步骤呢?当遇到片段扩增失败,或载体难构建的情况,也许你会不停优化引物设计、或更换试剂,你是否知道可以通过序列优化增加载体构建成功率呢?
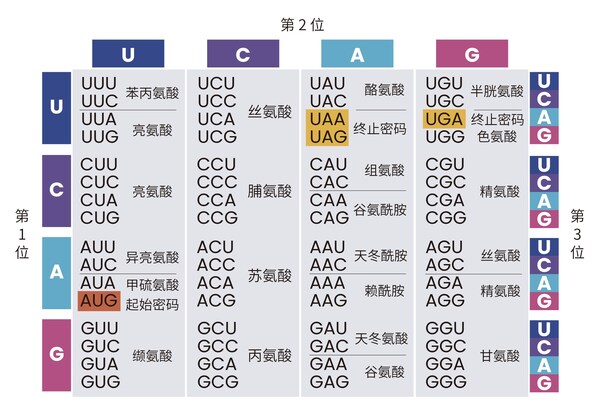
在理解密码子优化的重要性之前,需先了解密码子在基因表达中的作用。DNA序列通过转录和翻译产生蛋白质,而每个密码子由三个核苷酸组成,指定一个特定的氨基酸。这种对应关系在密码子表中明确列出,例如,密码子"ATG"编码甲硫氨酸(Met),开始蛋白质的合成。苯丙氨酸(Phe)对应的密码子有UUU和UUC,而终止密码子包括UAA、UAG和UGA。这种精确的编码方式确保了蛋白质合成的准确性和特异性。
虽然不同的密码子可编码相同的氨基酸,但不同生物体对密码子的使用存在偏好。这种偏好与每个生物体内特定tRNA的丰度相关。基因表达水平与密码子偏好性之间存在强相关性,同一氨基酸在不同宿主中,其对应的密码子会表现出不同程度的偏好[1]。因此,直接使用未优化的基因序列可能导致外源基因在宿主,特别是异源宿主中的表达效率低下。
密码子优化(codon optimization)是在不改变基因编码蛋白质的前提下,通过调整基因编码序列中的密码子使用频率,使用物种偏好密码子并避免稀有密码子得到优化序列,来提高外源基因在宿主细胞中表达水平的技术[2]。
对于自己构建载体的客户来说,是无法进行密码子优化的,因此采用基因合成方法来构建载体就成为了首选。
序列优化涉及更广泛的序列调整,包括优化GC含量、避免重复序列、消除不利的mRNA二级结构等,目的是提高整个基因的稳定性和表达效率,同时不改变最终编码的氨基酸序列。
- 优化原则
(1)密码子偏好性
密码子适应指数CAI是指异源mRNA序列中密码子和宿主细胞最佳密码子使用频率相符合的程度,理论上此值越接近1,外源mRNA在宿主细胞中的蛋白表达越高,通常CAI<0.8时,被认为需要进行密码子优化。使用宿主细胞中使用频率高的同义密码子替换外源mRNA序列中的密码子,例如,在大肠杆菌中,某些密码子如TAA和TAG可能很少使用,而GCA(编码丙氨酸)的使用频率较高。且避免出现稀有密码子,稀有密码子可能导致翻译停顿,增加翻译时的错误率,从而降低蛋白质产量。同时考虑高频和次高频的密码子组合。
注意CAI值过高可能导致外源基因在宿主细胞中过度表达,进而使得蛋白质在合成过程中积累过快,无法正确折叠,从而形成包涵体。包涵体是指在细胞内形成的不溶性蛋白质聚集物,通常是因为蛋白质未能正确折叠或是由于蛋白质表达水平过高。适当的密码子优化可以帮助调控蛋白质的表达速率,减少包涵体的形成,提高目标蛋白的正确折叠和可溶性表达。
(2)GC含量
理想的GC含量通常在40%-60%之间。过高的GC含量可能导致mRNA二级结构过于稳定,过低则可能影响转录效率。
(3)mRNA二级结构
复杂和稳定的二级结构会影响翻译效率,特别是序列中有和核糖体结合位点或翻译起始位点互作的序列,会阻止翻译的进行,合理优化翻译起始区的mRNA二级结构,可提高蛋白表达水平。
- 免费密码子及序列优化
擎科生物独有的 GeneOptimizer 软件密码子优化算法是不仅局限于密码子层面的序列深度优化技术。支持上万种宿主优化和定制化服务。优化的主要参数包括:密码子偏好性、GC 含量、减少poly(A) 结构、重复序列、核糖体结合位点、酶切位点等。在保证密码子适应指数CAI和GC含量处在合理范围内,尽可能减少其他负面因素的影响,求得帕累托最优解,进而完成对基因表达的优化。擎科生物可为基因合成客户提供免费的密码子及序列优化服务。
通过自动化序列分析评估软件在不到一次眨眼的时间就能完成一个准确的序列优化(0.1s/个)。
- 自动化方案设计
擎科生物自主开发的自动化方案设计软件,根据载体和序列特点可批量设计多种高效构建方案,提高载体构建成功率。
案例分享:
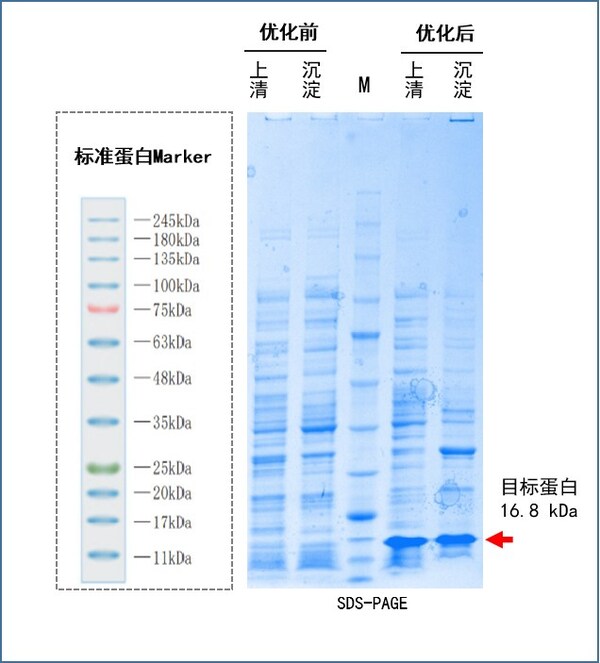
(1)目标蛋白16.8 kDa,密码子优化前蛋白不表达,密码子优化后蛋白大量表达。
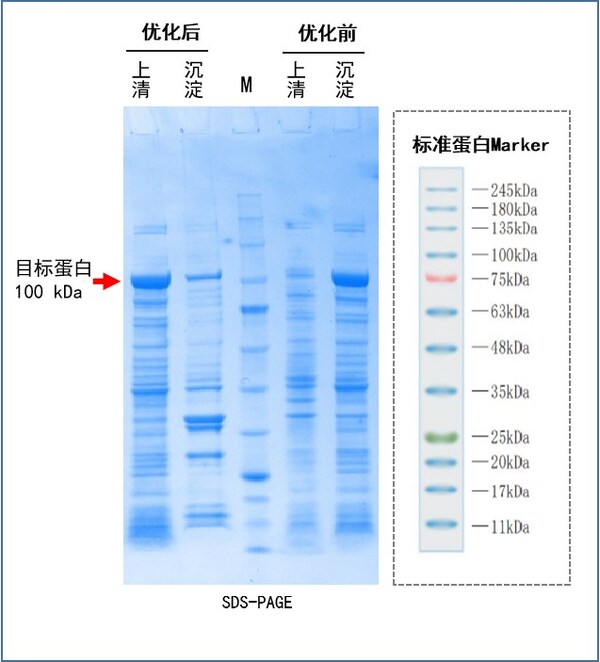
(2)目标蛋白100 kDa,密码子优化前包涵体表达,密码子优化后上清可溶表达75%。
[1] Sharp, P. M., & Li, W. H. (1987). "The Codon Adaptation Index—a measure of directional synonymous codon usage bias, and its potential applications." Nucleic Acids Research, 15(3), 1281-1295.
[2] Gustafsson, C., Govindarajan, S., & Minshull, J. (2004). "Codon bias and heterologous protein expression." Trends in Biotechnology, 22(7), 346-353.
- 中国人寿财险怀化市中心支公司 “3.15”系列活动之进商圈
- 《180天重启计划》研讨会举办:都市亲情题材的艺术突破与社会镜鉴
- 翁虹联合国教科文演讲:做自己,取悦自己,绽放女性魅力
- “智汇湾区 科技春韵” ——香港大学中国商业学院2025新春发展论坛暨广州校友会春茗圆满举行
- 昱能科技连续6年获EUPD顶级认证,携光储方案闪耀荷兰光伏展!
- 三优生物共同轻链抗体库,开启双抗药物新篇章
- 深化产教融合 助力就业新路径 兰州城市学院与网易集团共育数字文娱人才
- 兰州理工大学与阿里国际数字商业集团产教融合研讨会成功召开
- "极目3·15"共筑满意消费新生态,Witsbb健敏思以“无敏”营养再掀品质消费浪潮
- 廖校贤主任——中西医结合,打造肿瘤治疗新模式